近日,必威Betway东盟体育于彬教授团队在生物信息学研究领域取得新进展,相关成果以“DeepUTF: Locating transcription factor binding sites via interpretable dual-channel encoder-decoder structure”为题发表在人工智能领域的顶级期刊Pattern Recognition (中科院一区TOP期刊,影响因子7.5)。于彬教授为论文的唯一通讯作者,2021级研究生丁鹏举为第一作者,必威BETWAY官网为第一完成单位。

转录因子 (Transcription Factors, TFs) 通过与DNA序列上特定区域结合,参与调控基因表达,在顺式调控过程中发挥关键作用。这些特定区域包含高度保守的核苷酸序列,被称为转录因子结合位点 (Transcription Factor Binding Sites, TFBSs)。TFBSs的布局模式通常被称为调控元件或顺式调控motifs,代表特定核苷酸序列模式在基因组中的排列方式。精准地识别TFBSs及其对应的motifs对于深入理解基因调控网络以及表征基因组的特定功能特征至关重要,有助于解决当下生物医学领域的一些难题。近年来,尽管针对TF-DNA结合的特异性研究涌现了许多计算工具和数据库,但大部分方法过于依赖已知模式或先验信息,导致其在处理未知结合位点时的性能下降。现有的一些方法需要大量计算资源,限制了其在大规模基因组数据上的应用,预测性能仍有进一步提升的空间。此外,对TF-DNA结合机制的系统性理解和特征学习过程的可解释性仍然存在挑战,使得研究者难以深入理解这些方法的生物学基础。因此,建立一种可解释的人工智能预测模型,以准确定位TFBSs并预测TF-DNA结合motifs,已经成为生物信息学和合成生物学研究领域的一项关键挑战。
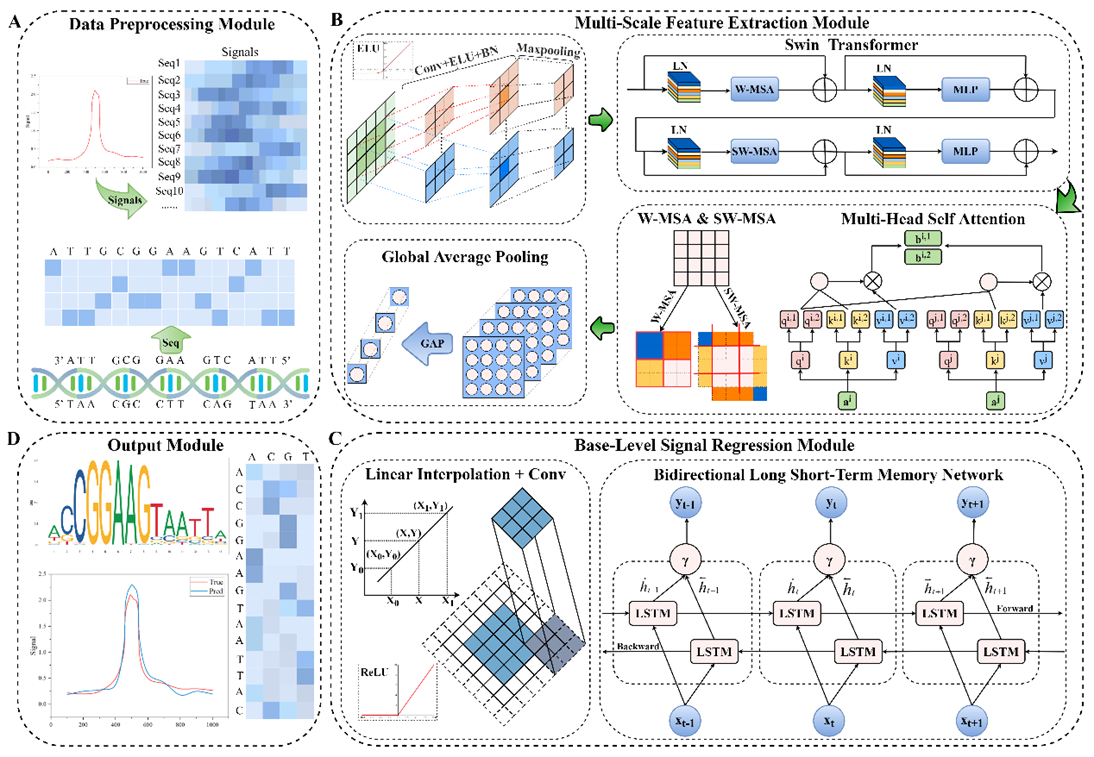
本研究提出一种新颖的可解释性双通道Encoder-Decoder架构DeepUTF,它接收DNA序列作为输入,综合应用改进的U-Net架构、Swin Transformer结构和并行Bi-LSTM在碱基级别上对转录因子结合区域的信号进行建模,从而完成序列分类、准确定位TFBSs和TF-DNA结合motifs预测任务。本研究解释了Swin Transformer结构在捕获多种依赖关系和聚焦学习重要特征方面的效用,对模型的输出以及对TF-DNA结合motifs的预测结果进行了可解释性分析,深入阐述了模型的内在机制和特征学习过程。在53个ChIP-seq数据集上的实验表明,DeepUTF在预测TFBSs方面明显优于几种最先进的算法,训练好的模型可以准确预测TF-DNA结合motifs,并且可以寻找间接的TF-DNA结合motifs。此外,本文将预测结果与PDB数据库进行对比,证明DeepUTF模型预测结果具有较好的连续性和准确性,为深入挖掘基因调控网络提供了有力支持。
文章链接:https://doi.org/10.1016/j.patcog.2024.111279
此外,该团队近一年来基于转录因子结合位点预测在生物信息学领域的高水平SCI期刊Briefings in Bioinformatics取得两项重要科研成果,已发表论文:
DeepSTF: predicting transcription factor binding sites by interpretable deep neural networks combining sequence and shape. Briefings in Bioinformatics, 2023, 24(4): bbad231. (IF=9.5, TOP CCF B)。于彬教授为论文的唯一通讯作者,研究生丁鹏举为第一作者。
Cooperation of local features and global representations by a dual-branch network for transcription factor binding sites prediction. Briefings in Bioinformatics, 2023, 24(2): bbad036. (IF=9.5, TOP CCF B)。于彬教授、张法教授为论文的通讯作者,本科生余雨桐为第一作者。
以上研究成果得到了国家自然科学基金面上项目、山东省自然科学基金面上项目的资助。